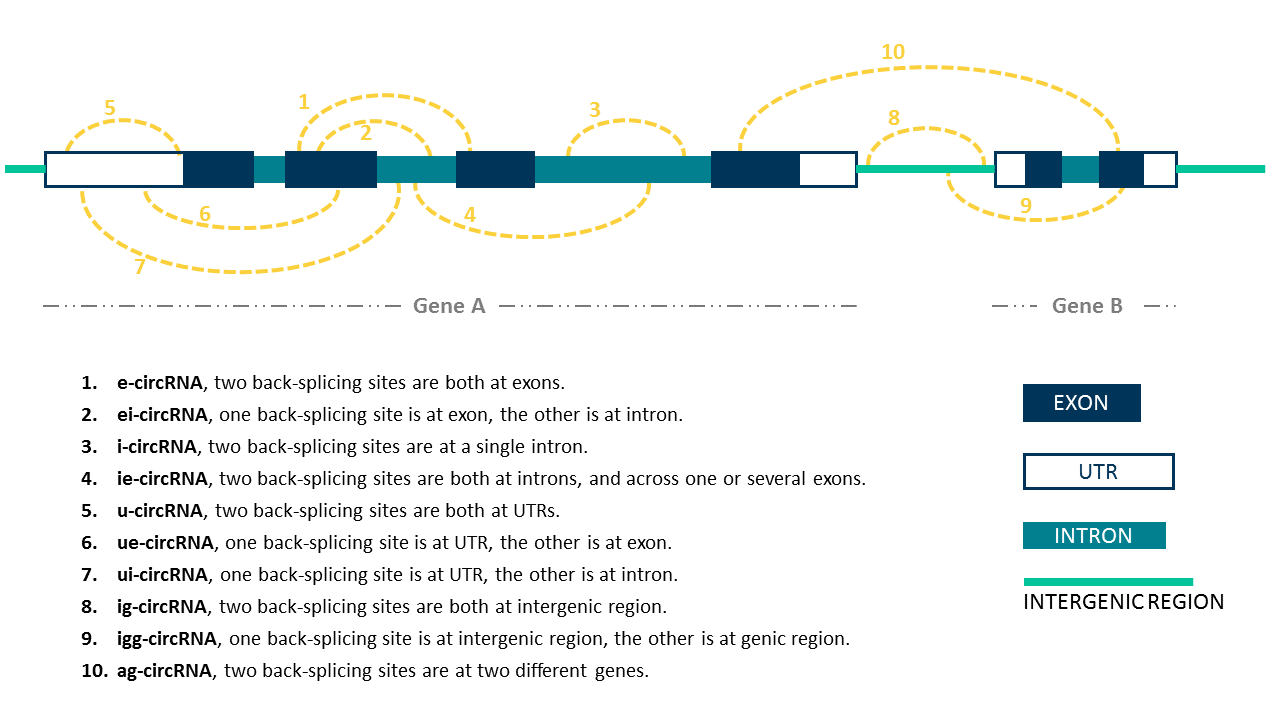
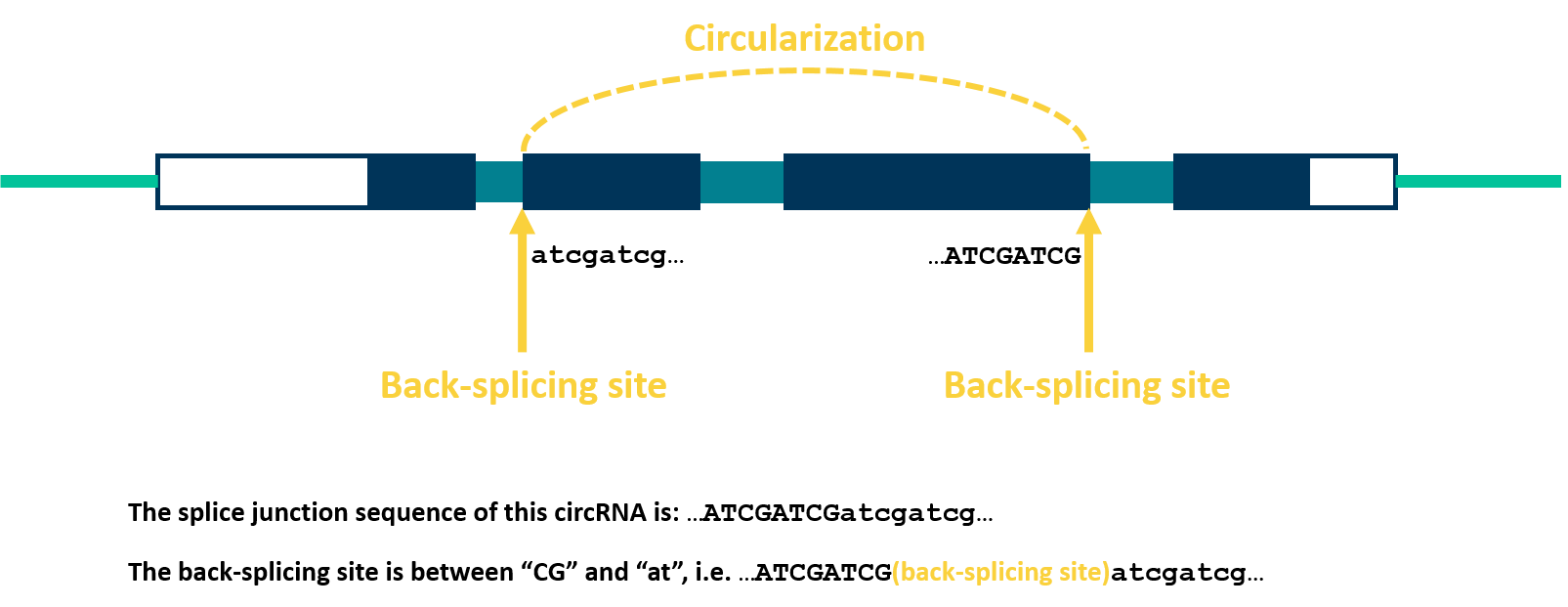
circRNA
Detailed infomation of each circRNA
Detail information of Mt_circ_ig.6
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.560399377 |
| Functional Information | |
|---|---|
| Coding potential | NA |
| Potential coding position |
NA |
| Potential amino acid sequence |
NA |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | NA |
| Other Information | |
|---|---|
| References | Philips et al., 2020 |