circRNA
Detailed infomation of each circRNA
Detail information of Zm00001d041889_circ_g.2
| General Information | |
|---|---|
| CircRNA Name | Zm00001d041889_circ_g.2 |
| ID in PlantcircBase | zma_circ_007658 |
| Alias | Zm03circ00049 |
| Organism | Zea mays |
| Position | chr3: 142436633-142438737 JBrowse» |
| Reference genome | AGPv4.38 |
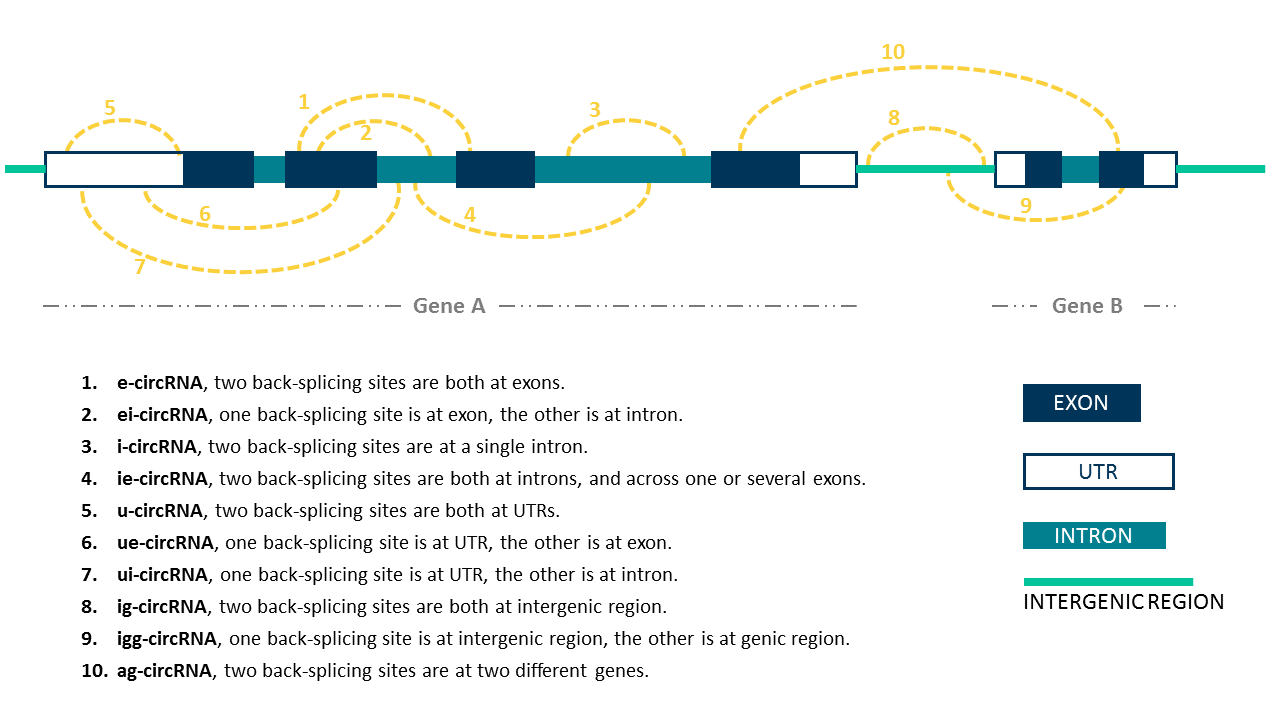
| Type | u-circRNA |
| Identification method | CIRI2 |
| Parent gene | Zm00001d041889 |
| Parent gene annotation |
protein_coding |
| Parent gene strand | + |
| Alternative splicing | Zm00001d041889_circ_g.1 Zm00001d041889_circ_g.3 Zm00001d041889_circ_g.4 Zm00001d041889_circ_g.5 Zm00001d041889_circ_g.6 Zm00001d041889_circ_g.7 Zm00001d041889_circ_g.8 Zm00001d041889_circ_g.9 Zm00001d041889_circ_g.10 Zm00001d041889_circ_g.11 Zm00001d041889_circ_g.12 Zm00001d041889_circ_g.13 Zm00001d041889_circ_g.14 Zm00001d041889_circ_g.15 Zm00001d041889_circ_g.16 Zm00001d041889_circ_g.17 Zm00001d041889_circ_g.18 Zm00001d041889_circ_g.19 |
| Support reads | NA |
| Tissues | leaf, endosperm |
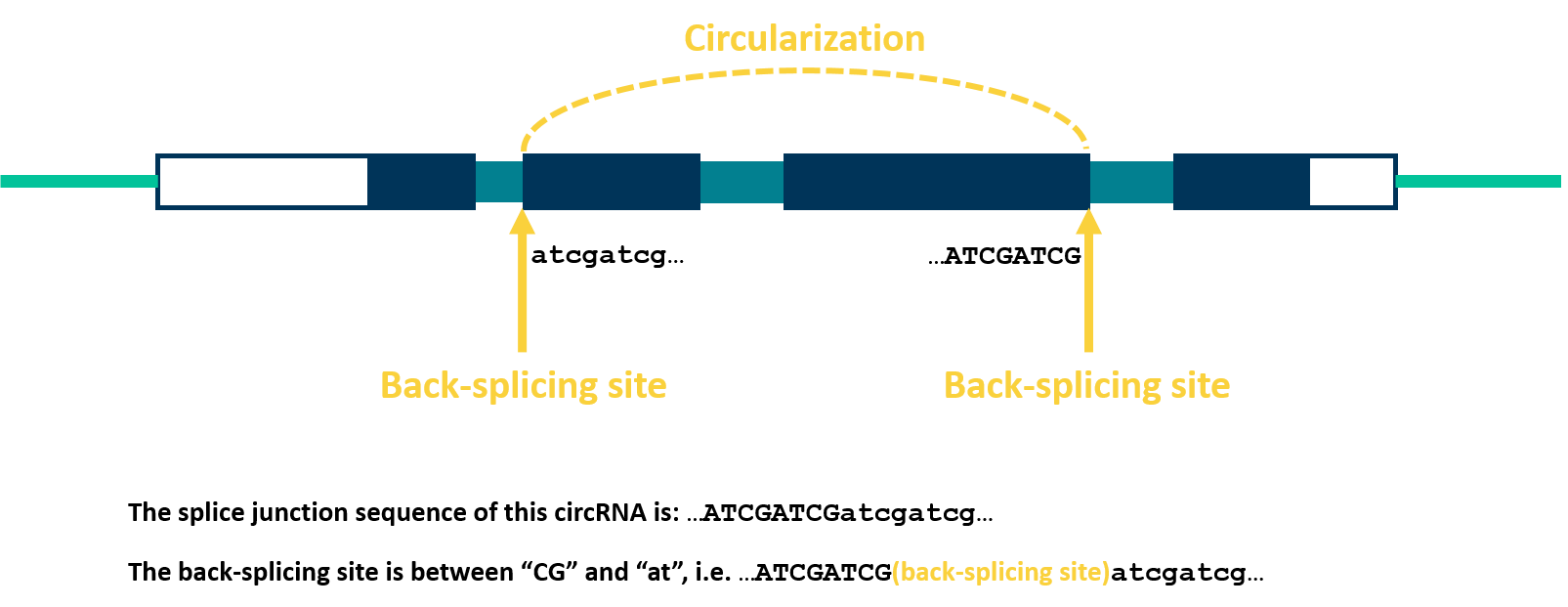
| Exon boundary | Yes-Yes |
| Splicing signals | GT-AG |
| Number of exons covered | Zm00001d041889_T075:5 Zm00001d041889_T114:6 Zm00001d041889_T014:7 Zm00001d041889_T025:7 Zm00001d041889_T034:5 Zm00001d041889_T096:7 Zm00001d041889_T001:6 Zm00001d041889_T138:4 Zm00001d041889_T027:7 Zm00001d041889_T144:2 Zm00001d041889_T065:6 Zm00001d041889_T018:7 Zm00001d041889_T131:7 Zm00001d041889_T097:7 Zm00001d041889_T081:7 Zm00001d041889_T089:7 Zm00001d041889_T059:7 Zm00001d041889_T108:6 Zm00001d041889_T007:7 Zm00001d041889_T066:7 Zm00001d041889_T143:2 Zm00001d041889_T054:7 Zm00001d041889_T129:7 Zm00001d041889_T033:7 Zm00001d041889_T136:4 Zm00001d041889_T003:6 Zm00001d041889_T057:6 Zm00001d041889_T051:6 Zm00001d041889_T022:6 Zm00001d041889_T132:6 Zm00001d041889_T145:3 Zm00001d041889_T101:5 Zm00001d041889_T064:7 Zm00001d041889_T117:7 Zm00001d041889_T041:7 Zm00001d041889_T099:5 Zm00001d041889_T113:7 Zm00001d041889_T002:7 Zm00001d041889_T103:7 Zm00001d041889_T070:6 Zm00001d041889_T107:6 Zm00001d041889_T134:7 Zm00001d041889_T035:7 Zm00001d041889_T019:7 Zm00001d041889_T039:5 Zm00001d041889_T031:4 Zm00001d041889_T005:6 Zm00001d041889_T048:7 Zm00001d041889_T079:7 Zm00001d041889_T012:7 Zm00001d041889_T016:7 Zm00001d041889_T106:7 Zm00001d041889_T042:7 Zm00001d041889_T036:7 Zm00001d041889_T061:7 Zm00001d041889_T087:7 Zm00001d041889_T045:4 Zm00001d041889_T142:2 Zm00001d041889_T073:7 Zm00001d041889_T139:4 Zm00001d041889_T116:5 Zm00001d041889_T135:6 Zm00001d041889_T092:7 Zm00001d041889_T137:4 Zm00001d041889_T094:7 Zm00001d041889_T077:6 Zm00001d041889_T141:3 Zm00001d041889_T140:1 Zm00001d041889_T062:6 Zm00001d041889_T029:5 Zm00001d041889_T102:4 Zm00001d041889_T021:5 Zm00001d041889_T068:6 Zm00001d041889_T083:7 Zm00001d041889_T010:4 Zm00001d041889_T082:7 Zm00001d041889_T115:6 Zm00001d041889_T085:5 Zm00001d041889_T049:6 Zm00001d041889_T090:5 Zm00001d041889_T086:6 Zm00001d041889_T047:6 |
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.460488003 |
| Functional Information | |
|---|---|
| Coding potential | Y |
| Potential coding position |
142436669-142438673(-) |
| Potential amino acid sequence |
MLFNRENQRQNIFISLFSYSVIIMLQSFGVEIS*(-) |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | NA |
| Other Information | |
|---|---|
| References | Han et al., 2020 |