circRNA
Detailed infomation of each circRNA
Detail information of Zm00001d025734_circ_g.1
| General Information | |
|---|---|
| CircRNA Name | Zm00001d025734_circ_g.1 |
| ID in PlantcircBase | zma_circ_010322 |
| Alias | zma_circ_0000056 |
| Organism | Zea mays |
| Position | chr10: 127909873-127913484 JBrowse» |
| Reference genome | AGPv4.38 |
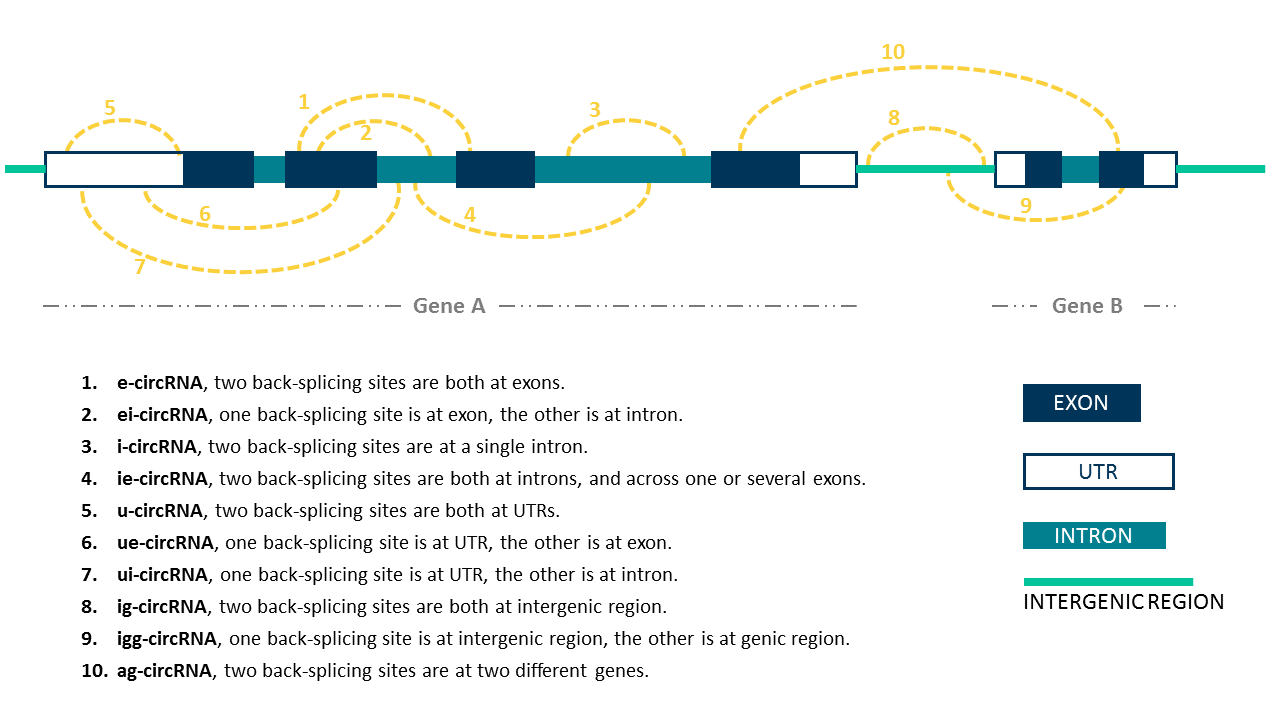
| Type | u-circRNA |
| Identification method | find_circ |
| Parent gene | Zm00001d025734 |
| Parent gene annotation |
protein_coding |
| Parent gene strand | + |
| Alternative splicing | NA |
| Support reads | NA |
| Tissues | leaf, root |
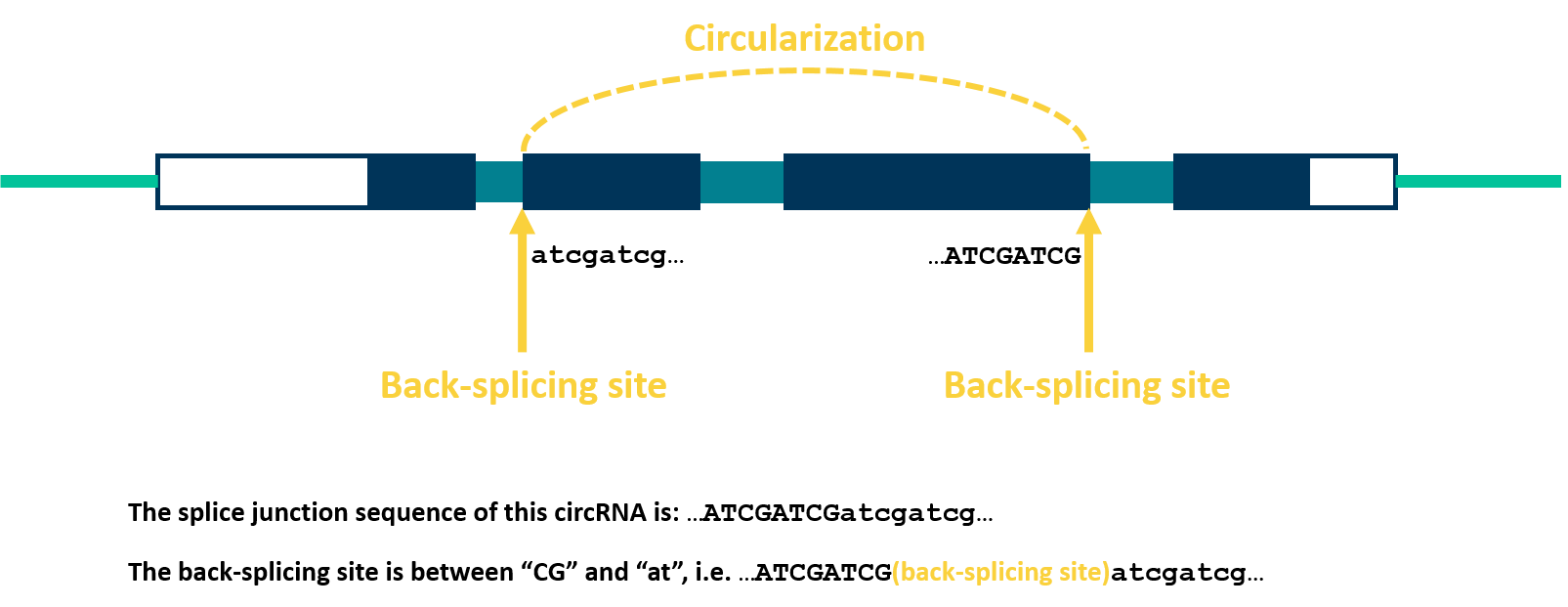
| Exon boundary | Yes-Yes |
| Splicing signals | GT-AG |
| Number of exons covered | Zm00001d025734_T011:7 Zm00001d025734_T012:7 Zm00001d025734_T001:6 Zm00001d025734_T017:4 Zm00001d025734_T016:6 Zm00001d025734_T007:7 Zm00001d025734_T010:7 Zm00001d025734_T009:6 Zm00001d025734_T002:6 Zm00001d025734_T013:7 Zm00001d025734_T005:7 Zm00001d025734_T015:7 Zm00001d025734_T014:7 Zm00001d025734_T003:7 Zm00001d025734_T008:7 Zm00001d025734_T006:6 |
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.113622972 |
| Functional Information | |
|---|---|
| Coding potential | Y |
| Potential coding position |
127913438-127910085(+) 127910383-127909873(+) 127909929-127913479(-) |
| Potential amino acid sequence |
MQRMCFYYGRFGWTSKVRYKRWLWWTRFGMAVGALQLVAAIYLMFVIVRDLSNERRSTSCFFGM LVAILSYLFPNFVHNFLQVLVCW*(+) MVYNLGQDKADRVSVRALIALFLILSWVVIIVQCFTGSDILRWRSFYATHDMAWKAHYREVFDH GIREALCCLGRAKYLTVLEEDEVYSVARLLGDLVAYRASGTGHLELLAGLALLQKHGNLSELQT DLVEASHELMQEAAFLHPFAEACYTGPLLDFGRNPILFPCAWVYRQGVLTPWARRRRPALDGDN WWRGHAAAFLRFVNIPPKALLKGRVCQSKREAAYFVVVLHDKRTVVIGVRGTETPEDLITDGLC RECAFTMEDLDGLVK*(+) MHQLPYQIWSTTTSVCTSLY*(-) |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | NA |
| Other Information | |
|---|---|
| References | Ma et al., 2021b |