circRNA
Detailed infomation of each circRNA
Detail information of Os08g0451000_circ_g.1
| General Information | |
|---|---|
| CircRNA Name | Os08g0451000_circ_g.1 |
| ID in PlantcircBase | osa_circ_037251 |
| Alias | NA |
| Organism | Oryza sativa |
| Position | chr8: 22014862-22017674 JBrowse» |
| Reference genome | IRGSP-1.0.38 |
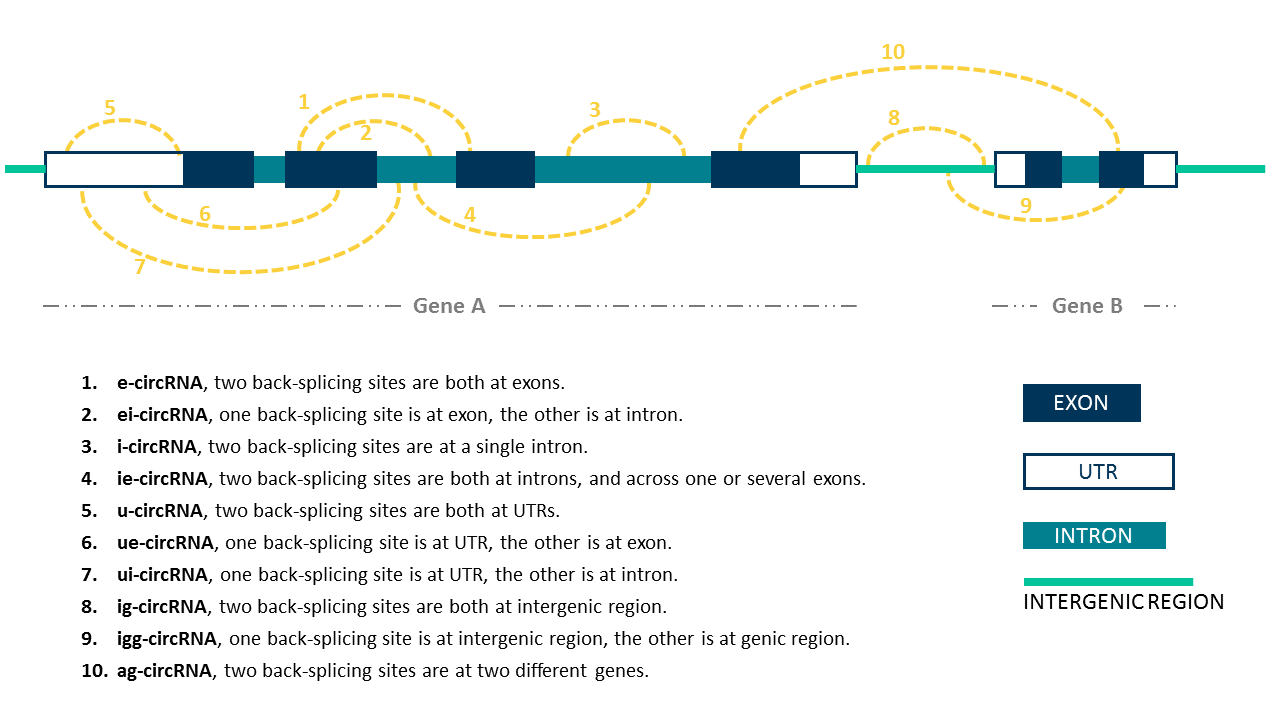
| Type | e-circRNA |
| Identification method | CIRCexplorer |
| Parent gene | Os08g0451000 |
| Parent gene annotation |
Sec34-like protein family protein. (Os08t0451000-01) |
| Parent gene strand | + |
| Alternative splicing | Os08g0451000_circ_g.2 |
| Support reads | 1 |
| Tissues | root |
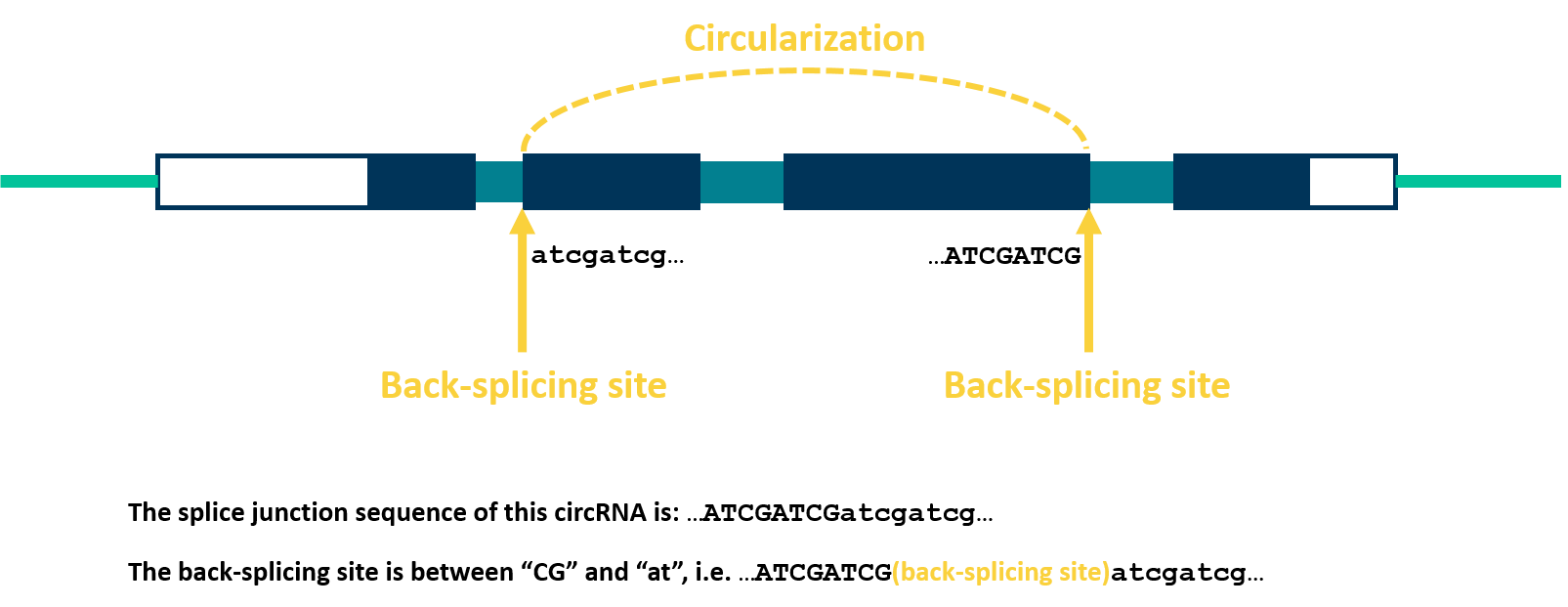
| Exon boundary | Yes-Yes |
| Splicing signals | GT-AG |
| Number of exons covered | Os08t0451000-01:9 |
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.108418761 |
| Functional Information | |
|---|---|
| Coding potential | Y |
| Potential coding position |
22014932-22017671(+) 22017462-22014871(+) 22014918-22017669(-) |
| Potential amino acid sequence |
MDAVLVNTHQFYKWFSELESAMKSETEEKYRLYESTLEERVKTCDGILQQVDDTLNLFEELQSL HLSVATKTKTLHDACDQLLLEKQRLIEFAEALRSRLNYFDELENVSTSFYSQTMNIGNEQFLPL LKRLDDCISYVENNPQYAESAVYLVKFRQLQSRALGMIRSHVLSILKAASSQVQAAIRGNGSAK NVVTEGVEASLIYVRFKAAASEEKSLVKDGGVVVPEKEAALEEGAMDAVLVNTHQFYKWFSELE SAMKSETEEKYRLYESTLEERVKTCDGILQQVDDTLNLFEELQSLHLSVATKTKTLHDACDQLL LEKQRLIEFAEALRSRLNYFDELENVSTSFYSQTMNIGNEQFLPLLKRLDDCISYVENNPQYAE SAVYLVKFRQLQSRALGMIRSHVLSILKAASSQVQAAIRGNGSAKNVVTEGVEASLIYVRFKAA ASEEKSLVKDGGVVVPEKEAALEEGAMDAVLVNTHQFYKWFSELESAMKSETEEKYRLYESTLE ERVKTCDGILQQVDDTLNLFEELQSLHLSVATKTKTLHDACDQLLLEKQRLIEFAEALRSRLNY FDELENVSTSFYSQTMNIGNEQFLPLLKRLDDCISYVENNPQYAESAVYLVKFRQLQSRALGMI RSHVLSILKAASSQVQAAIRGNGSAKNVVTEGVEASLIYVRFKAAASEEKSLVKDGGVVVPEKE AALEEGAMDAVLVNTHQFYKWFSELESAMKSETEEKYRLYESTLEERVKTCDGILQQVDDTLNL FEELQSLHLSVATKTKTLHDACDQLLLEKQRLIEFAEALRSRLNYFDELENVSTSFYSQTMNIG NEQFLPLLKRLDDCISYVENNPQYAESAVYLVKFRQLQSRALGMIRSHVLSILKAASSQVQAAI RGNGSAKNVVTEGVEASLIYVRFKAAASE(+) MSCQYLKPLHPRFKRRFEVMVQQKMLLQRVWKHPLSMFASRQLLVRRKV*(+) MQPLFLAQPHPHPSLNFSPH*(-) |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | NA |
| Other Information | |
|---|---|
| References | Chu et al., 2017 |