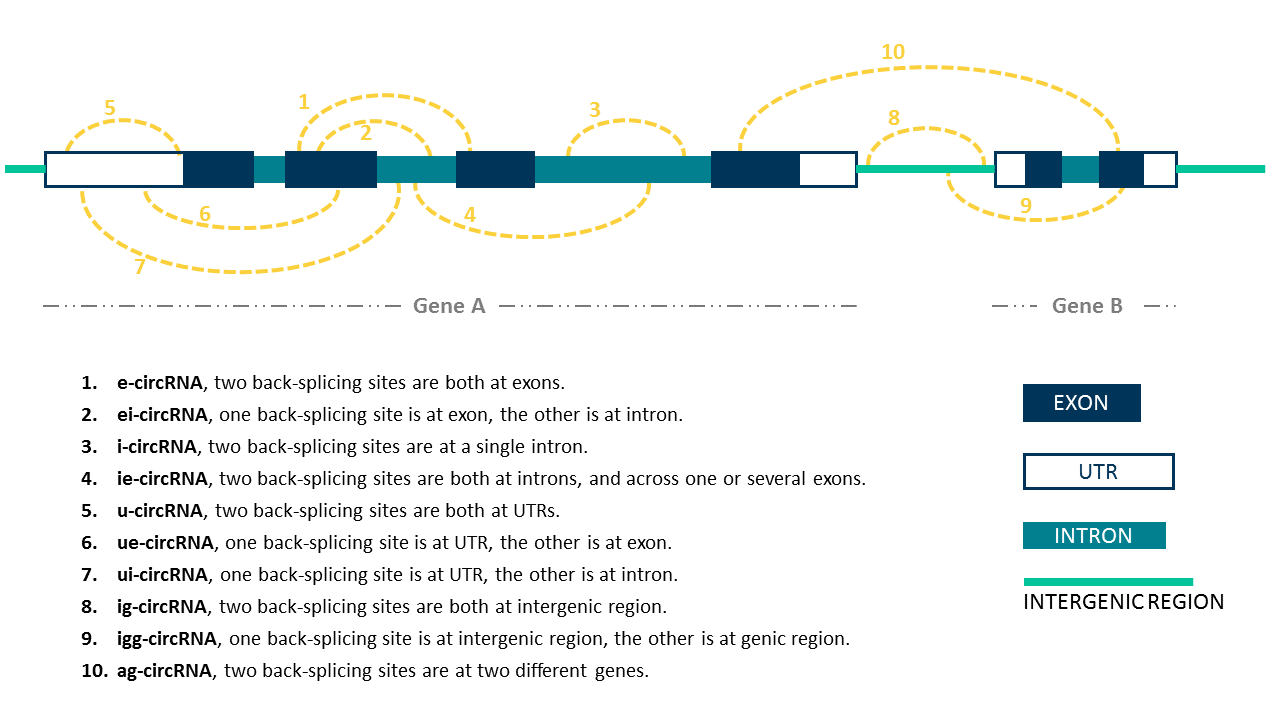
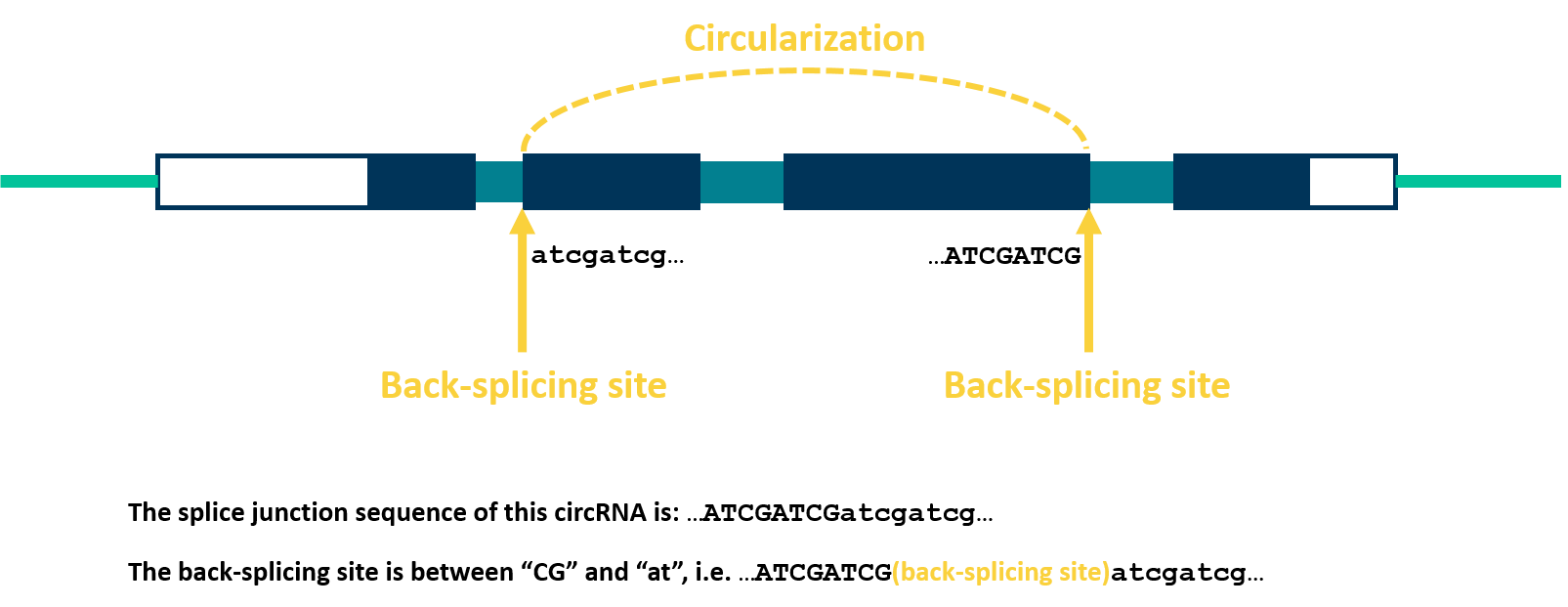
circRNA
Detailed infomation of each circRNA
Detail information of Zm00001d001978_circ_g.3
| General Information | |
|---|---|
| CircRNA Name | Zm00001d001978_circ_g.3 |
| ID in PlantcircBase | zma_circ_007020 |
| Alias | zma_circ_0001040, GRMZM2G017654_C2 |
| Organism | Zea mays |
| Position | chr2: 3814970-3815553 JBrowse» |
| Reference genome | AGPv4.38 |
| Type | u-circRNA |
| Identification method | find_circ, CIRI2 |
| Parent gene | Zm00001d001978 |
| Parent gene annotation |
MAP3K epsilon protein kinase 1 |
| Parent gene strand | + |
| Alternative splicing | Zm00001d001978_circ_g.2 |
| Support reads | NA |
| Tissues | leaf, root |
| Exon boundary | Yes-Yes |
| Splicing signals | GT-AG |
| Number of exons covered | Zm00001d001978_T022:2 Zm00001d001978_T007:2 Zm00001d001978_T103:2 Zm00001d001978_T030:2 Zm00001d001978_T014:2 Zm00001d001978_T107:2 Zm00001d001978_T005:2 Zm00001d001978_T130:2 Zm00001d001978_T096:2 Zm00001d001978_T031:2 Zm00001d001978_T111:2 Zm00001d001978_T008:2 Zm00001d001978_T009:2 Zm00001d001978_T089:2 Zm00001d001978_T117:2 Zm00001d001978_T037:2 Zm00001d001978_T012:2 Zm00001d001978_T122:2 Zm00001d001978_T074:2 Zm00001d001978_T033:2 Zm00001d001978_T039:2 Zm00001d001978_T002:2 Zm00001d001978_T018:2 Zm00001d001978_T084:2 Zm00001d001978_T020:2 Zm00001d001978_T011:2 Zm00001d001978_T026:2 Zm00001d001978_T077:2 Zm00001d001978_T088:2 Zm00001d001978_T042:2 Zm00001d001978_T101:2 Zm00001d001978_T016:2 Zm00001d001978_T080:2 Zm00001d001978_T118:2 Zm00001d001978_T120:2 Zm00001d001978_T079:2 Zm00001d001978_T104:2 Zm00001d001978_T133:2 Zm00001d001978_T017:2 Zm00001d001978_T060:2 Zm00001d001978_T114:2 Zm00001d001978_T058:2 Zm00001d001978_T015:2 Zm00001d001978_T019:2 Zm00001d001978_T087:2 Zm00001d001978_T081:2 Zm00001d001978_T075:2 Zm00001d001978_T013:2 Zm00001d001978_T109:2 Zm00001d001978_T121:2 Zm00001d001978_T125:2 Zm00001d001978_T073:2 Zm00001d001978_T115:2 Zm00001d001978_T131:2 Zm00001d001978_T071:2 Zm00001d001978_T006:2 Zm00001d001978_T124:2 Zm00001d001978_T028:2 Zm00001d001978_T091:2 Zm00001d001978_T055:2 Zm00001d001978_T049:2 Zm00001d001978_T076:2 Zm00001d001978_T010:2 Zm00001d001978_T090:2 |
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.132150161 |
| Functional Information | |
|---|---|
| Coding potential | Y |
| Potential coding position |
3815234-3814984(+) 3815502-3814984(+) |
| Potential amino acid sequence |
MDSAAQSKSEELHDGNLELTEGISSNNVALVKDNVVLNKDPTLVLHEKLPVESSFGDADLNGKV MAHELLQGGLPSKIELENKESSTVEDGDVFSFQAGRQNIDFQMEYRRG*(+) MVMYSHFRLEDRILTSKWNIDGDDESSRGDNNSGFCDTPGDTRATIASNVDQVNGRNGPNMDSA AQSKSEELHDGNLELTEGISSNNVALVKDNVVLNKDPTLVLHEKLPVESSFGDADLNGKVMAHE LLQGGLPSKIELENKESSTVEDGDVFSFQAGRQNIDFQMEYRRG*(+) |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | responsive to drought stress |
| Other Information | |
|---|---|
| References | Ma et al., 2021b;Zhang et al., 2019 |