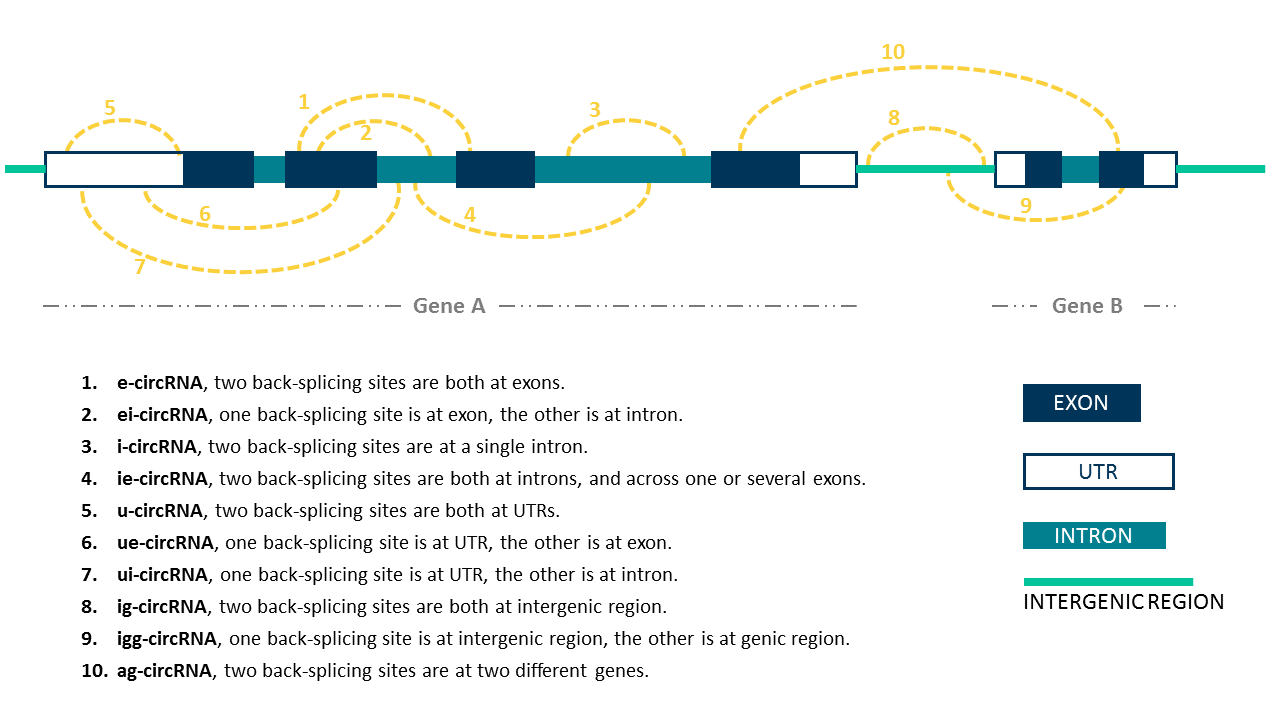
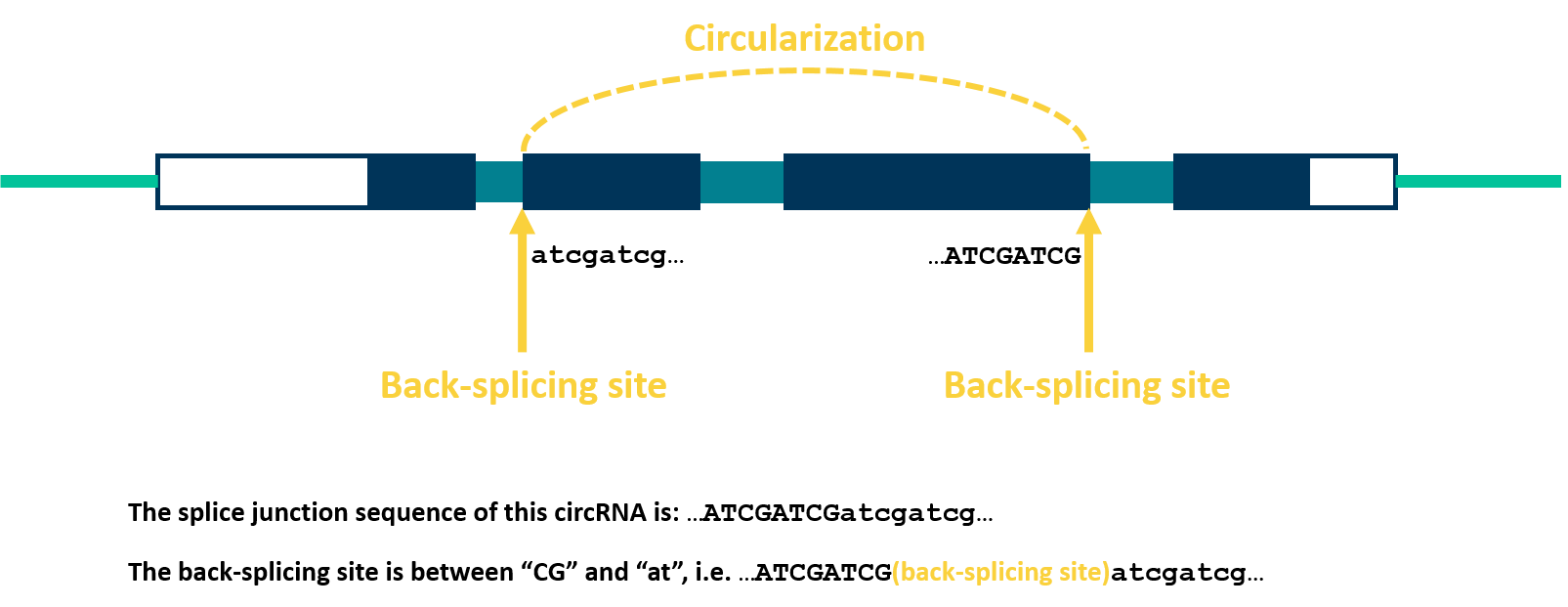
circRNA
Detailed infomation of each circRNA
Detail information of TGACv1_scaffold_030417_1BL_circ_ig.1
| General Information | |
|---|---|
| CircRNA Name | TGACv1_scaffold_030417_1BL_circ_ig.1 |
| ID in PlantcircBase | tae_circ_000082 |
| Alias | circRNA1094 |
| Organism | Triticum aestivum |
| Position | chrTGACv1_scaffold_030417_1BL: 51652-54385 JBrowse» |
| Reference genome | TGACv1.38 |
| Type | ig-circRNA |
| Identification method | find_circ |
| Parent gene | NA |
| Parent gene annotation | NA |
| Parent gene strand | NA |
| Alternative splicing | TGACv1_scaffold_030417_1BL_circ_ig.2 |
| Support reads | NA |
| Tissues | root |
| Exon boundary | NA-NA |
| Splicing signals | GT-CA |
| Number of exons covered | NA |
| Conservation Information | |
|---|---|
| Conserved circRNAs | NA |
| PMCS | 0.108158772 |
| Functional Information | |
|---|---|
| Coding potential | N |
| Potential coding position |
NA |
| Potential amino acid sequence |
NA |
| Sponge-miRNAs | NA |
| circRNA-miRNA-mRNA network | VISUALIZATION |
| Potential function description | NA |
| Other Information | |
|---|---|
| References | Ren et al., 2018 |